荧光流式细胞实验检测细胞周期并使用Python绘图
实验原理
荧光流式细胞术是根据荧光信号分离单细胞的技术,其识别的荧光可以是活细胞内表达的,也可以是固定细胞上体外标记的。因为在细胞分裂过程中遗传物种会翻倍,可以使用DNA染料染色固定细胞的方法观察细胞周期。其通常使用乙醇固定、PI染色(有可以使用DAPI)后即可上机流式细胞仪,得到.fcs文件。
无论流式筛选还是流式检测,其实验成功最最关键的点是对荧光信号的准确识别,需要通过多个通道的信号参数,成功过滤掉细胞碎片、粘连体细胞以及阴性细胞。需要注意的是,这一数据分析过程是在上机时需要即刻判断的。
流式数据的展示和简单分析其实可以有很多种软件直接打开并进行处理,但受制于收费版权、或缺少一些关键参数,这里探索了一个使用Python文件读取并分析流式数据的方案。
一点活细胞流式筛选的经验
-
细胞上机前一定需要过滤网,滤网除了正下面,侧面也可以使用,滤网可以洗干净后泡酒精、高压蒸汽灭菌循环使用
-
细胞消化后,不要用太多培养基重悬,流式仪器的分选速度很高,但如果细胞悬液过稀,分选速度就提不上去
-
在分选阳性细胞的时候,最好带一个同类型的阴性细胞,找一下荧光信号的位置,否则在阳性率很低的时候容易错过真正的信号
-
不要过早准备细胞悬液,流式上机前务必将细胞从管底部弹起
-
活细胞分选96孔板时,外围一圈可以只加PBS而不分选细胞,因为在培养箱中很容易干,细胞生长状态会明显差
流式的通道和参数
流式通道主要可以分为散射光通道和荧光通道。荧光通道和荧光显微镜是一致的,散射光信号通常测定两种方向的,即FSC(前向角散射)和SSC(侧向角散射)
-
FSC的值代表细胞的大小:细胞体积越大,其FSC值就越大。所以可以利用细胞的FSC值初步比较细胞的大小,利用FSC值对细胞进行分群和分类
-
SSC的值代表细胞的颗粒度:细胞越不规则,细胞表面的突起越多,细胞内能够引起激光散射的细胞器或者颗粒性物质越多,其SSC值就越大
-
所以可以利用细胞的SSC值初步比较细胞的颗粒度,利用SSC值对细胞进行分群和分类
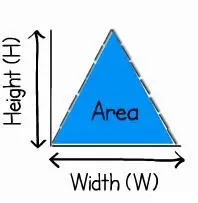
关于流式参数:每个细胞通过激光的时候,机器都会记录一个波状信号(一个拱形的信号)
-
H就是这个拱的高度(代表的是信号的强度)
-
A就是这个拱的曲线下面积,代表信号强度和细胞大小的关系
-
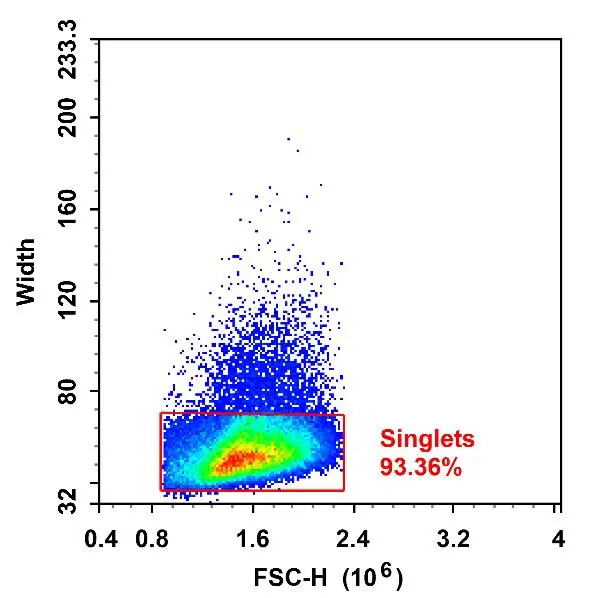
参数W,是width的缩写,是这个拱的宽度,代表细胞通过激光束所花的时间
关于粘连体
-
粘连体,是指两个或更多个细胞粘在一起(大多数是两个)的现象
-
单个细胞进入激光照射区到离开,电脉冲信号由0到达峰值,再降回0,被记录为一个峰值
-
当细胞与细胞会粘在一起,连续通过激光照射区,也会被仪器认定为一次信号
-
当两个细胞挤在一起通过仪器,与单个细胞相比,测量高度不变,宽度和面积变为两倍,即出现FSC-H不变,FSC-A和W增大的现象
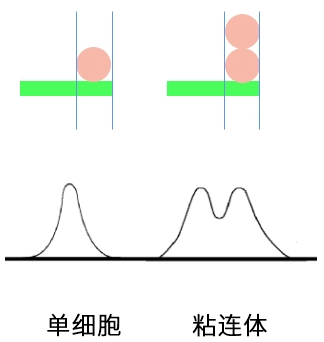
在分选GFP阳性细胞时,可能会有5种情况:单个荧光阳性、单个荧光阴性,粘连双阳、粘连双阴、粘连一阴一阳
-
如果不去出粘连体,一阴一阳的细胞会通过GFP阳性的检测,从而影响分选效率
-
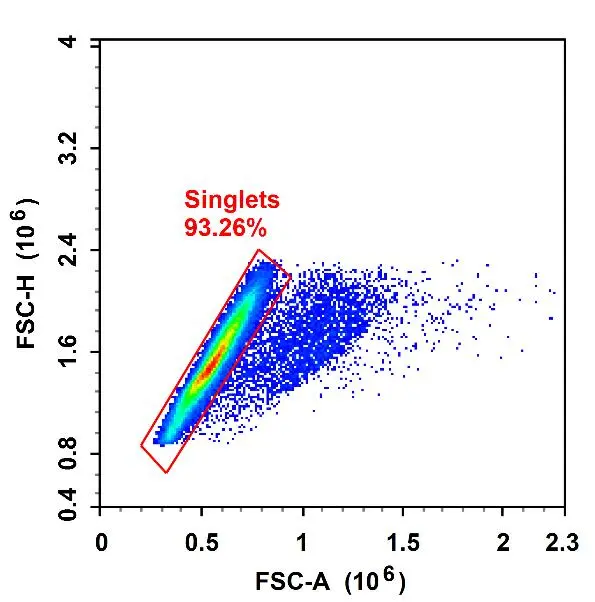
可通过细胞的FSC-A(Area)与FSC-H(Height)参数排除掉粘连体
-
由于双粘连体的脉冲宽度大于单个细胞脉冲的宽度,基本可以达到两倍的关系,所以也可以使用FSC-H与W来排除粘连细胞
-
但需要注意的是,如果检测样本中各种细胞的大小差异较大,此种分析方式可能不太适用
虽然流式在逻辑上可以完美排除掉粘连体,避免粘连体造成的假阳性影响。但是,如果粘连体太多,即使在分析数据时排除掉,对实验结果的影响仍然很大,对仪器管路也不利。因此在前期样本制备时预防粘连体,才是首选之策!
使用cytoflow模块分析流式数据
1、引包,需要注意的是cytoflow需要python3.8.5或3.8.12,高版本或低版本或5-12中间都无法成功安装
import cytoflow as flow
from matplotlib import pyplot as plt
plt.style.use('default')
import seaborn as sns
import numpy as np
from scipy.stats import gaussian_kde2、使用cytoflow模块读取.fcs数据,并标记实验条件,得到整合后的数据data为pandas的Dataframe,可以使用类似data[data['cycle']=='WT']的命令筛选需要的数据。本次实验使用双Thyminde同步化的方法得到各个时期的细胞(G2R是在双Thyminde处理的基础上配合CDK1抑制剂RO3306得到的G2期细胞)
tube1 = flow.Tube(file = 'Data001.fcs', conditions = {'cycle' : 'WT'})
tube2 = flow.Tube(file='Data002.fcs', conditions = {'cycle' : 'G1/S'})
tube3 = flow.Tube(file='Data003.fcs', conditions = {'cycle' : 'G2'})
tube4 = flow.Tube(file='Data004.fcs', conditions = {'cycle' : 'G2R'})
import_op = flow.ImportOp(conditions = {'cycle' : 'str'}, tubes = [tube1, tube2, tube3, tube4])
ex = import_op.apply()
data = ex.data3、可以使用matplotlib等方法可视化观察这个Dataframe;在cytoflow中提供了一个Histogram Plot的方法
hist = flow.HistogramView()
hist.channel = 'FL2-H'
hist.plot(ex)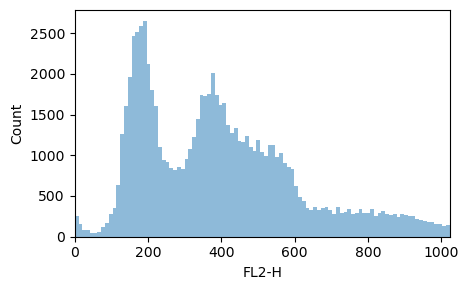
4、为了能好看一点,这里就不用seaborn的密度图了,自己定义一个根据密度改变颜色的散点图
def colorscatter(ax, data, x, y, cmap='jet'):
x, y = data[x], data[y]
xy = np.vstack([x,y])
z = gaussian_kde(xy)(xy)
idx = z.argsort()
x, y, z = x[idx], y[idx], z[idx]
r = ax.scatter(x, y, c=z, s=5, cmap=cmap)
ax.set(xlabel='FSC-H', ylabel='SSC-H')
return r5、画图;这里绘制时选择了H参数,是希望能够谨慎处理粘连体,在一些文章和软件中是使用了A参数
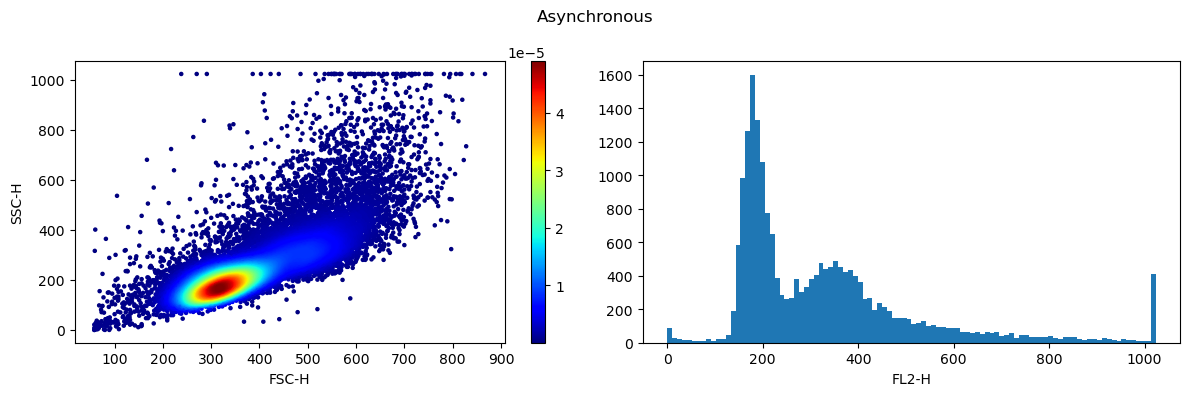
fig, axes = plt.subplots(1, 2, figsize=(12, 4))
cs = colorscatter(axes[0], data[data['cycle']=='WT'], x='FSC-H', y='SSC-H')
fig.colorbar(cs, ax=axes[0])
axes[1].hist(data[data['cycle']=='WT']['FL2-H'], bins=100)
axes[1].set_xlabel('FL2-H')
fig.suptitle('Asynchronous')
plt.tight_layout()
plt.show()