Venn和Upset Diagram的绘制
对于Venn图,一直没有找到好看的Python工具,因而我一直在使用网页工具来完成:
本次任务中,是希望利用已经公开发布的Apex2质谱数据,在完全不相关的实验中,找到一些共有的因子,从而建立BlackList。网页工具数据集有点少,并且对数据挖掘不够,这里就自己尝试了一些韦恩图工具。任务前期已经通过数据收集和整理,构建了表格apex2col.csv,表格中每一列是一个数据集,一共8列,需要找到并可视化这8个数据集的交叉情况
使用Python绘制韦恩图
引包和数据整理
import pandas as pd
from itertools import islice
from venn import venn
from venn import pseudovenn
data = pd.read_csv('../Rscript/apex2col.csv',encoding='gbk')
x = data.to_dict(orient='list')
x = {k: set([i for i in v if not pd.isna(i)]) for k,v in x.items()}绘图
pseudovenn(dict(islice(x.items(),6)), cmap="plasma", hint_hidden=False, legend_loc="best")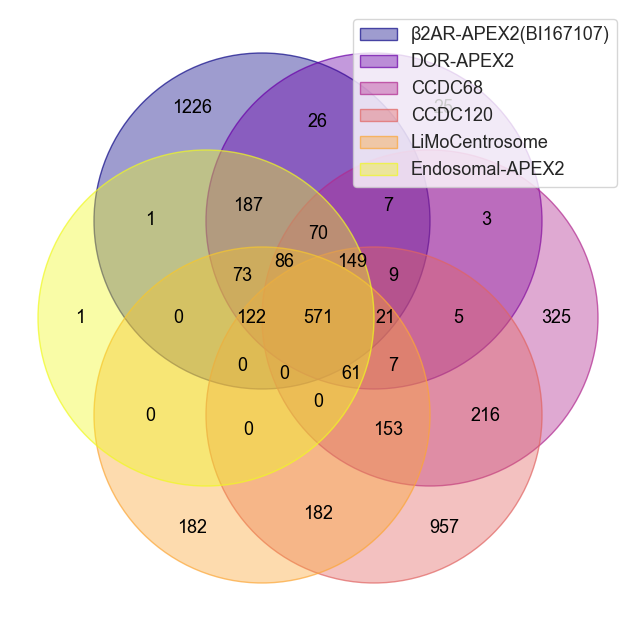
venn(dict(islice(x.items(),5)), legend_loc="best")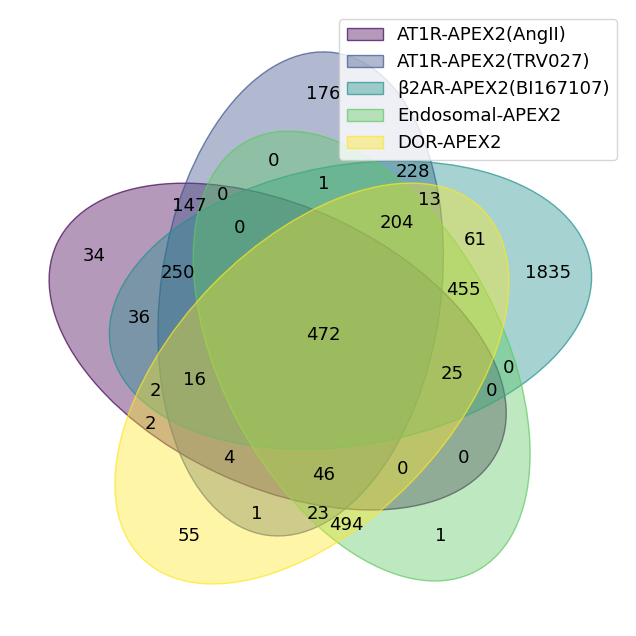
使JS工具绘制交互式Upset Diagram
Upset Diagram是多组数据相互比较的一种柱状图,能够清楚地看到数据集之间的交集。
这个Github仓库提供了一个可交互的JS绘图工具,很好用,很漂亮。但是它要求提供零一值的表格,并使用Json格式提供数据集基本信息和绘图配置,下面的Python代码是将原csv格式文件转化为目标数据格式的脚本。
data = pd.read_csv('../Rscript/apex2col.csv', encoding='gbk')
x = data.to_dict(orient='list')
x = {k: set([i for i in v if not pd.isna(i)]) for k,v in x.items()}
genes = np.unique([j for i in list(x.values()) for j in i])
res = []
for i in genes:
row=[i]
for j in x.values():
row.append(1) if i in j else row.append(0)
res.append(row)
colname = ['gene'] + list(x.keys())
resdata = pd.DataFrame(np.array(res), columns=colname)
resdata.to_csv('Apex2colUpsetDigarmData.csv', index=False, encoding='gbk')将如下文件上传至服务器并获取原数据获取链接(我这里直接放到了一个github仓库中),并将其按照格式填充进如下的json文件中,并加载进该工具
PS:似乎不能将数据放在本地,必须使用网络的url;但是绘图的程序代码可以直接下载到本地,然后将json数据加载进程序就可以
{
"file": "https://raw.githubusercontent.com/l392537594/proteinanno/main/XXX.csv",
"name": "Test",
"header": 0,
"separator": ",",
"skip": 0,
"meta": [
{ "type": "gene_id", "index": 0, "name": "Gene_ID" }
],
"sets": [
{ "format": "binary", "start": 1, "end": 7 }
]
}这个是最后的效果,很有利于进一步的数据挖掘
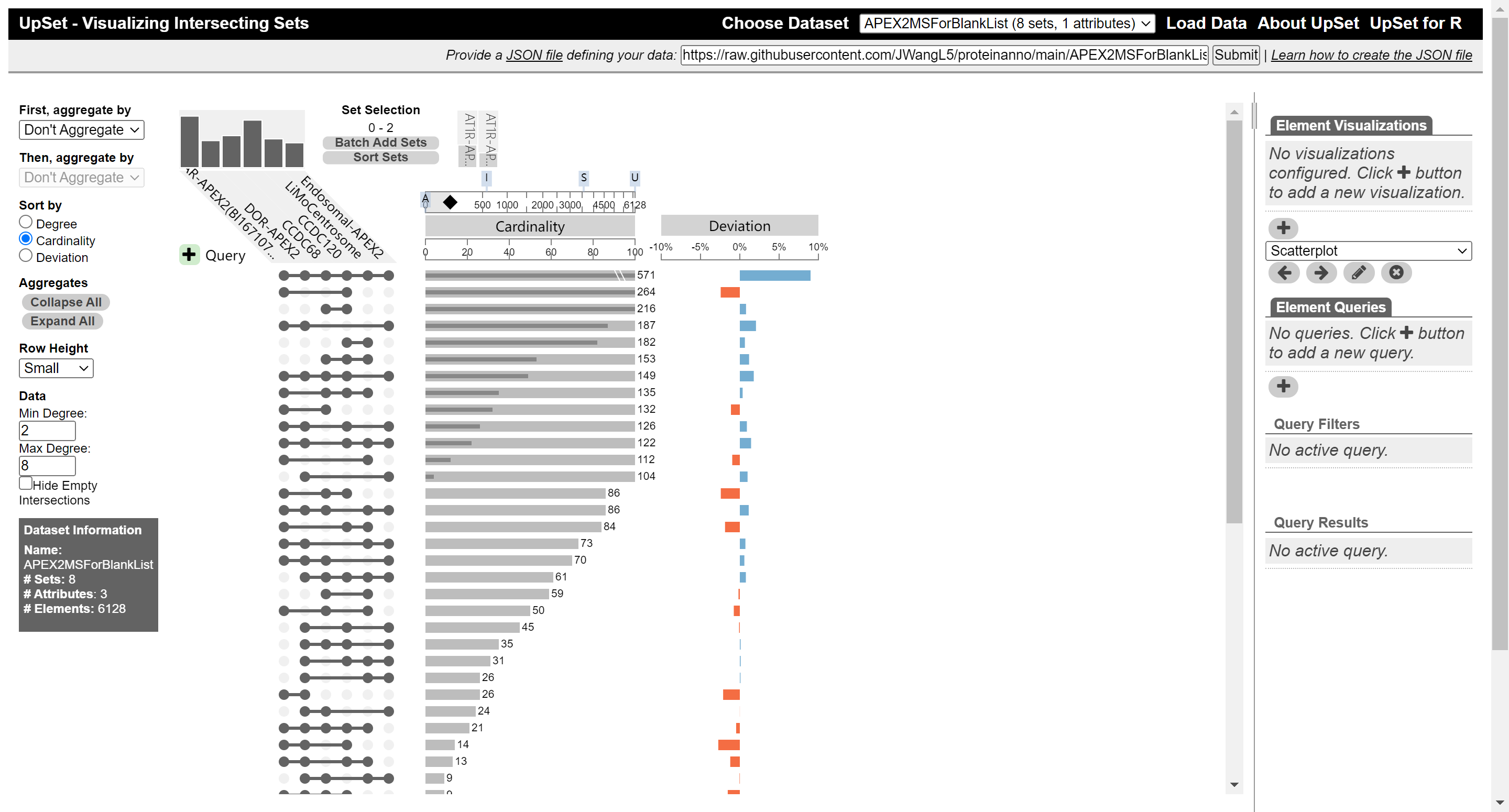
使用R绘制韦恩图
效果很一般,并且同时绘制的数据组个数有限
install.packages("VennDiagram")
library(VennDiagram)
a <- read.csv("Rscript/apex2col.csv",
sep = ',',
header = T,
na.strings = "")
A <- as.list(na.omit(a$AT1R.APEX2.AngII.))
B <- as.list(na.omit(a$AT1R.APEX2.TRV027.))
C <- as.list(na.omit(a$`β2AR.APEX2.BI167107.`))
D <- as.list(na.omit(a$Endosomal.APEX2))
E <- as.list(na.omit(a$DOR.APEX2))
F <- c(as.list(na.omit(a$CCDC68)))
G <- c(as.list(na.omit(a$CCDC120)))
venn.diagram(x=list(F,G),
filename = 'Rscript/tets2.png',
category.names = c("F","G"))